▶ 服务介绍
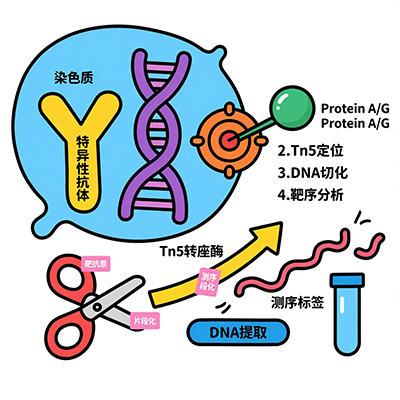
甲基化RNA免疫共沉淀(MeRIP/m6A-seq)是通过特异性抗体富集含m6A修饰的RNA片段,结合高通量测序或qPCR分析转录组-wide甲基化分布的技术。利用抗m6A抗体与随机打断的RNA片段孵育,沉淀修饰片段后进行下游检测(测序/qPCR)。
▶ 实验步骤
1. Total RNA样品检测
在进行MeRIP实验之前,首先需要对RNA样品进行检测以确保其质量和数量满足后续实验的要求:
琼脂糖凝胶电泳分析:检测RNA的降解程度及是否存在污染,确保具有明显的18S或28S主带且条带清晰。
Qubit2.0精确量化:对RNA浓度进行精确测量,总RNA检测总量不低于10μg。
Agilent2100完整性检测:检测RNA的完整性,RIN值不低于7.5。
2. RNA片段化
取10μg总RNA,加入RNA片段化试剂(如Invitrogen提供的试剂),在70℃下反应10分钟,将RNA打断成约100nt的片段。随后使用乙醇法将片段化的RNA沉淀。
3. m6A富集
这是MeRIP实验的核心步骤之一,旨在通过抗体特异性结合m6A修饰的RNA片段来富集目标RNA:
将含有Protein A和Protein G的磁珠用IP缓冲液(如150 mM NaCl, 10 mM Tris-HCl pH 7.5)洗涤。
与5μg m6A抗体(如Millipore提供的抗体)在4℃下孵育2小时。用IP缓冲液洗涤磁珠两次,再用IP缓冲液重悬磁珠,加入片段化的RNA,在4℃下翻转孵育4小时。
用IP缓冲液在4℃下洗涤磁珠三次,再用m6A竞争性洗脱液在4℃下孵育1小时。
收集含有洗脱m6A RNA的上清至新的试管中,并使用苯酚:氯仿:3-甲基丁醇(125:24:1)进行纯化。
4. 文库构建
使用SMARTer® Stranded Total RNA-seq Kit v2-Pico Input Mammalian User Manual根据说明书对IP与Input样品进行反转录及文库构建。利用AMPure XP bead进行片段大小选择,获得最终文库。
5. 文库质检
文库构建完成后,需要对其进行质量检测:
使用Qubit2.0进行初步定量,稀释文库至1ng/μl。
使用Agilent2100对文库的插入片段大小进行检测,确保插入片段大小符合预期。
使用qPCR方法对文库的有效浓度进行准确定量(文库有效浓度应为2nM),以确保文库质量。
6. 上机测序
文库检测合格后,根据不同文库的有效浓度及目标下机数据量的需求进行混合,并在Illumina Nova平台测序,测序策略为PE150。
7. 信息分析流程
原始下机数据质控:对测序数据进行初步处理,去除低质量读段。
比对参考基因组:将高质量读段比对到参考基因组上。
peak calling:识别m6A修饰的热点区域。
功能注释:对识别出的m6A修饰区域进行功能注释,分析其可能参与的生物学过程